英国基因专家巴卢教授:确定病毒进入人类宿主,发生在去年年底
一项来自伦敦大学学院(UCL)最新研究显示:新冠病毒在去年10月至11月期间就首次感染了人类,随后在全球传播中表现出基因的多样性。新冠病毒在欧洲和美国开始传播的时间,比官方公布首例病例至少要早一个月。
这项研究结果于5月5日晚以预审的方式,在线发表在《感染,遗传学和进化》科学杂志网站上。通讯作者为英国基因专家、伦敦大学学院基因研究所主任弗朗索瓦·巴卢教授。

巴卢教授团队分析了GISAID平台上截至2020年4月19日、来自全球的7666名新冠患者的全基因组序列后发现:
01
新冠病毒在2019年10月6日至2019年12月11日期间首次从宿主感染人类;
02
感染人类后,病毒在首例感染后快速地扩散,迅速在世界范围内传播;
03
新冠病毒基因组中的198个位点已经发生了反复的独立突变,但这并不意味着它正在恶化,目前还不能确定变异是否改变病毒的传播性和毒性;
04
突变后的新冠病毒正在不断适应其人类宿主。
“我们非常、非常、非常有信心地确定病毒进入人类宿主,发生在去年年底。”巴卢教授表示。
01 新冠病毒去年10月至11月首次感染人类
研究小组通过分析7666名新冠患者的病毒基因组,确定了SARS-CoV-2病毒基因组的多样性特征。监测其积累和模式有可能为药物和疫苗开发提供信息。该项研究确定了新冠病毒中近200个复发性基因突变,“根据对公共基因组装配的大规模分析,SARS-CoV-2基因组中的198个位点似乎已经发生了反复的独立突变,可能表明病毒在不断适应其新宿主人类。”研究人员发现,SARS-CoV-2的全球遗传多样性中,有很大一部分是在受灾最严重的国家发现的,这表明病毒流行初期就开始了广泛的全球传播,而且大多数国家中没有单一的“零号患者”。
在包括英国在内,许多国家采样病毒的多样性几乎与全世界一样多,这意味着该病毒是独立多次进入英国的,而不是通过任何一个指数案例。研究人员从系统发生学估计,新冠大流行的开始时间可能在2019年10月6日至2019年12月11日左右之间,这也对应于自然宿主“跃入”人类的时间。该病毒在2019年底出现,然后迅速传播到全球。“这一研究排除了新冠病毒在发现以前可能早已开始传播、并已感染了大部分人口的假设。”巴卢教授表示,“所有病毒都会自然变异。突变本身不是一件坏事,没有任何迹象表明SARS-CoV-2的突变比预期快或慢。到目前为止,我们还不能说SARS-CoV-2是否或多或少变得更有致命性和传染性。”

值得注意的是,这一结论与此前一篇由复旦大学公共卫生临床中心张永振教授与合作者悉尼大学爱德华·霍尔莫斯(Edward Holmes)教授3月26日在Cell上发表文章的结论不同。
张永振和霍尔莫斯教授的论文称,新冠疫情在大规模暴发前,可能已经在人群中经历了一段时间的“隐性”传播,早期由于感染者无症状、症状轻微或者零星的肺炎病例未引起注意,直到病毒获得关键点位的突变,更好地适应人类宿主。
但巴卢教授坚信,他的研究结果排除了任何有关“病毒很早就在人群中传播并因此感染了大部分人口”的场景。
“我非常确定这个病毒是在去年晚些时候才感染人类的,因为我们收集的来自全球各个角落的所有基因序列样本,表现出的多个变异都是类似的。”巴卢教授表示。
研究人员称,识别出的微小遗传变化或突变未在病毒基因组中平均分配。由于基因组的某些部分几乎没有突变,那些不变部分可能是药物和疫苗开发的更好靶标。抵抗病毒的主要挑战是,如果病毒突变,疫苗或药物可能不再有效。“如果我们将精力集中在病毒中不太可能发生突变的部分上,就更有机会开发出长久有效的药物。”巴卢解释说,“我们需要开发出不轻易被病毒逃逸的药物和疫苗。”共同首席作者Lucy van Dorp博士(UCL遗传学研究所)补充说,“病毒之间的遗传差异或突变仍然很少。我们发现一些差异已经在大流行过程中多次出现,彼此独立发生。随着更多基因组的可用,我们需要继续监测这些差异,并研究确定它们的作用。”该研究小组开发了一种新的交互式,开放源代码在线应用程序,以便全球研究人员查看病毒基因组,并应用类似方法来了解其进化过程。Lucy van Dorp表示,“能在大流行的头几个月分析如此大量的病毒基因组,对于药物开发工作可能是无价的,同时也展示了过去十年中,基因组研究走了多远。全球数百名研究人员的巨大努力使我们都受益匪浅,他们一直在对病毒基因组进行测序并在线提供。”
02 SARS-CoV-2的变化
论文指出,SARS-CoV-2是一种可能为人畜共患病源的SARS样冠状病毒,于2019年12月在中国湖北省武汉市首次发现。此病毒已在全球传播,导致当前COVID-19大流行。第一个完整的基因组序列于2020年1月发布,自次已对数千个基因组进行了测序。该资源提供了对SARS-CoV-2过去人口统计资料的空前洞察力,还可以监视病毒是如何适应人类宿主的,为直接设计药物和疫苗提供信息。研究者策划了7666个公共基因组装配的数据集,并分析了随着时间的流逝基因组多样性的出现。结果与之前估计相符,并指出了到2019年底所有具有共同祖先的序列,在SARS-CoV-2跃入其人类宿主的时期就支持了这一点。由于广泛传播,该病毒在几个国家的遗传多样性概括了其全球遗传多样性的很大一部分。SARS-CoV-2基因组的区域迄今仍然基本不变,而其他区域已经积累了多样性。通过关注已独立多次出现的突变(同源性),研究者确定了SARS-CoV-2基因组中198个过滤后的复发突变。其中近80%在蛋白质水平上产生了非同义的变化,表明可能持续进行SARS-CoV-2的适应。来自病原体的基因序列数据分析日益被认为是传染病流行病学的重要工具。遗传序列数据揭示了关键的流行病学参数,例如暴发/流行的时间加倍,传播途径的重建以及可能来源和动物库的识别。此外,全基因组序列数据可以为药物和疫苗设计提供依据。基因组数据可用于鉴定与宿主相互作用的病原体基因,并能够表征病原体基因组的更多进化受限区域,应优先针对这些区域,以避免快速的药物和疫苗逃逸突变体。
结论一:SARS-CoV-2基因组多样性的出现
7666个SARS-CoV-2基因组为COVID-19大流行提供了清晰的地理和时间覆盖范围。其多样性表现为径向和线性布局中的最大似然系统发育。数据中有一个强大的时间信号,可通过7666个SARS-CoV-2的采样日期与“根到尖端”距离的统计显着相关性来捕获。具体而言,自最近分离的菌株与最新的共同祖先不同以来,它们在基因组中积累的突变量就超过了旧菌株。
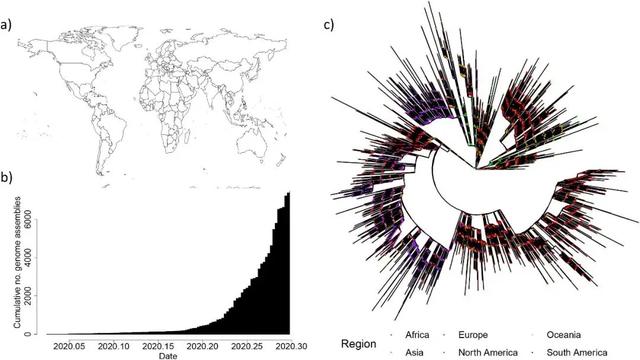
结论二:SARS-CoV-2无处不在
最大似然系统发育中的一些主要进化枝,主要由从同一大陆采样的菌株形成。但是,这可能表示时间信号而不是地理信号。最早的可用菌株是在亚洲收集的,COVID-19大流行始于亚洲,随后首先在欧洲,然后在美国进行了广泛的基因组测序工作。
在大多数国家中发现的SARS-CoV-2基因组多样性,从本质上概括了7666个基因组数据集中COVID-19的全球多样性。下图突出显示了在英国、美国、冰岛和中国发现的全球遗传多样性的比例,每个国家都有所有主要进化枝的代表。对于其他国家,例如澳大利亚,也是如此。
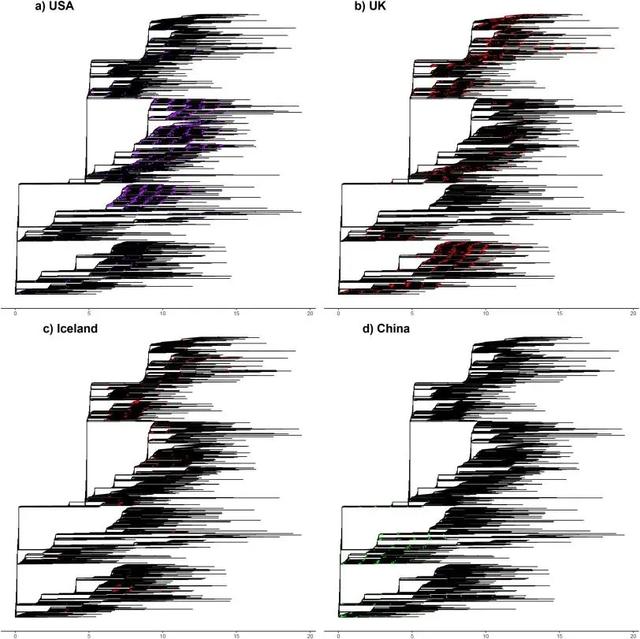
在不同国家流通的SARS-CoV-2种群的遗传多样性表明,这些地方每一个都是通过大量独立引入该病毒而播种的。但主要例外是中国,它是最初暴发的来源,那里仅存在全球多样性的一小部分。意大利在某种程度上也是如此,这是COVID-19大流行的早期焦点。
但是,这个全球数据集仅包含来自意大利的35个SARS-CoV-2基因组,因此流通中SARS-CoV-2菌株的某些遗传多样性可能仍未抽样。在多个国家概述的全球SARS-CoV-2人口的基因组多样性表明,COVID-19可能在全球范围内从大流行的早期开始广泛传播。
结论三:基因组比对和反复突变的遗传多样性
在整个比对中,遗传多样性存在差异,多态性经常出现在邻近的簇中。然而,由于缺乏随附的序列读取数据,并非所有位点都可以确认为真正的变异位置。但是,研究者仔细检查了在最大简约树构建步骤之后似乎多次出现的站点,鉴定出大量推定的同质性,将其过滤为198个位置的高置信度队列。流行传染病已经影响人类数千年了。仅在上个世纪,数种流行病就夺走了数百万人的生命,包括1957/58年的甲型H2N2流感大流行,第六次(1899–1923年)和第七次“埃尔托”霍乱大流行(1961-1975年),1981年至今的艾滋病大流行。COVID-19不受欢迎地提醒人们,传染病构成了死亡和破坏的主要威胁。与以前的大流行相比,当前形势的一个积极方面,是面对新冠肺炎时科学技术手段空前的可用性。特别是药物和疫苗的快速研发已经开始。现代药物和疫苗开发主要基于基因工程和分子水平上对宿主-病原体相互作用的理解。世界各地的科学家动员起来应对COVID-19大流行是非常了不起的。这包括全球科学界的壮举,在撰写本文时(2020年4月23日),该壮举已经产生并公开分享了超过11,000个完整的SARS-CoV-2基因组序列。这种空前的基因组资源为大流行提供了强有力的结论。例如,由多个独立小组进行的分析,将COVID-19大流行的起点定在2019年底。这排除了任何假定SARS-CoV-2可能早在被识别之前就已经流通并因此已经感染了很大一部分人口的情况。迄今为止,在人类传播的SARS-CoV-2中观察到的绝大多数突变可能是中性的,甚至是有害的。同质性,例如我们在此处检测到的同质性,可能是中性进化的产物或正在进行的选择的结果。在我们检测到的198个同质异体中,很可能是选择的真正目标





